Visualisierungen auf molekularer Ebene
 Der menschliche oder tierische Organismus ist eine der komplexesten Entwicklungen der Biologie. Viele chemische, physikalische und mechanische Vorgänge laufen täglich völlig autonom und ohne unser Zutun ab. Wie funktioniert die Nervweiterleitung, was passiert beim Zerlegen der Nahrung in ihre molekularen Bestandteile, wozu werden diese benötigt und warum werden wir krank? Viele Fragen interessieren uns schon im Kindesalter und bringen die Erwachsenen in Erklärungsschwierigkeiten.
Der menschliche oder tierische Organismus ist eine der komplexesten Entwicklungen der Biologie. Viele chemische, physikalische und mechanische Vorgänge laufen täglich völlig autonom und ohne unser Zutun ab. Wie funktioniert die Nervweiterleitung, was passiert beim Zerlegen der Nahrung in ihre molekularen Bestandteile, wozu werden diese benötigt und warum werden wir krank? Viele Fragen interessieren uns schon im Kindesalter und bringen die Erwachsenen in Erklärungsschwierigkeiten.
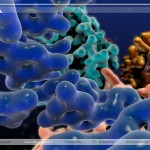
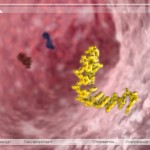
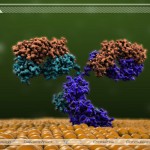
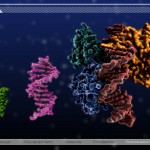
Selbst im hochwissenschaftlichen Bereich gibt es immer noch viele Dinge die wir nicht verstehen, die erst erforscht werden müssen und die selbst dann nur für fachkundige Menschen verständlich sind. Um derartige Vorgänge zu zeigen, die z. B. für das menschliche Auge normalerweise verborgen bleiben und um Zusammenhänge zu erklären die sonst nur durch komplexe Formeln, Gleichungen oder ausführliche Texte beschrieben werden können, eignen sich Computervisualisierungen besonders gut. Dazu haben wir in einer Machbarkeitsstudie mit Hilfe eines selbstgeschriebenen Programms PDB-Dateien ausgelesen und anschließend die molekulare Struktur der darin enthaltenen chemischen Verbindungen geometrisch rekonstruiert und graphisch sichtbar gemacht. Die hier gezeigten Arbeiten enstanden aus der Diplomarbeit „Hüllkurvenberechnung und -visualisierung molekularer Strukturen im dreidimensionalen Raum“ von Rainer Uhlemann. Der Unterschied zu anderen verfügbaren Programmen besteht darin, dass diese lediglich in der Lage sind Moleküle zu visualisieren, nicht aber um sie in 3D-Animationen einzubauen und somit in anderen Programmen nutzbar zu machen.